微生物痕量基因残留测定微滴数字 PCR 法
来源:武汉市灰藻生物科技有限公司 浏览量:569 发布时间:2024-11-25 16:45:32

前言
本文件按照 GB/T1.1-2020《标准化工作导则 第1部分:标准化文件的结构和起草规则》的规定起草。
请注意本文件的某些内容可能涉及专利。本文件的发布机构不承担识别专利的责任
本文件由中国标准化研究院提出并归口。
本文件起草单位:清华大学、中国科学院微生物研究所、河北省食品检验研究院、中国标准化研究院。
本文件主要起草人:张翀、邢新会、杜文斌、周巍、郭永、剪兴金、汪海燕、郑蒙、蔡东洋、马爱进
微生物痕量基因残留测定微滴数字 PCR 法
1范围
本文件规定了用微滴数字PCR法测定微生物痕量基因残留(100Pg/mL 以下)的方法。
本文件规定了用微滴数字PCR法测定微生物痕量基因残留(100Pg/mL 以下)的方法。
2规范性引用文件
下列文件中的内容通过文中的规范性引用而构成本文件必不可少的条款。其中,注日期的引用文件,仅该日期对应的版本适用于本文件;不注日期的引用文件,其最新版本(包括所有的修改单)适用于本文件。
GB/T6682分析实验室用水规格和试验方法
3术语和定义
下列术语和定义适用于本文件
3.1痕量基因残留trace gene residue
微生物加工产品在后续分离纯化中很难去除的痕量(含量在100Pg/ml以下)核酸
4原 理
利用液滴微流控技术,将待测生物样品所残留的目标核酸PCR扩增体系分割成几十份到几万份包含一个拷贝的核酸分子微升级油包水微滴。按照常规PCR体系对其进行扩增,通过荧光探针实现信号读出。所产生微滴阳性信号的数目即代表了原始样本中目标核酸分子的拷贝数,从而直接数出目标核酸分子的个数,对起始样品的痕量基因残留进行定量。
5试剂或材料
除非另有规定,仅使用分析纯试剂。
5.1水为GB/T6682规定的一级水。将一级水在121℃下高压加热灭菌15min制备得到无菌水。
5.2DNA 提取试剂盒。
5.3微滴数字 PCR 反应试剂盒。
5.4引物和探针
5.4.1酿酒酵母引物和探针:
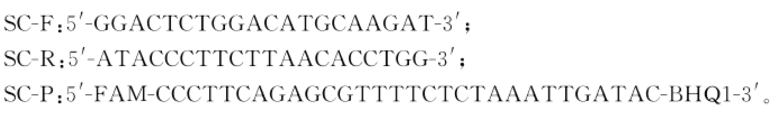
5.4.2 植物乳杆菌引物和探针:
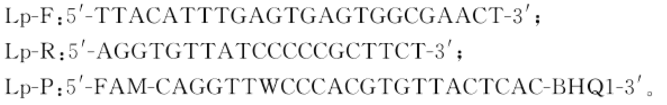
5.5酵母浸出粉胨葡萄糖培养基(YPD培养基):称取蛋白胨20.0g,酵母提取物10.0g,葡萄糖20.0g,先加人 990ml,水溶解,并调节pH至7.0~7.4的范围内,再定容至1000mL。然后在121℃下高压加热灭菌 15 min。
5.6 乳酸细菌培养基(MRS培养基):称取蛋白胨10.0g,牛肉粉10.0g,酵母粉5.0g,葡萄糖20.0g,柠檬酸三铵 2.0 g,乙酸钠5.0g,磷酸氢二钾 2.0g,硫酸镁0.2g,硫酸锰 0.05g,加人吐温-801.0ml,先加人990ml水溶解,并调节pH至6.2~6.6的范围内,再定容至1000ml。然后在121 ℃下高压加热灭菌 15 min。
5.7酿酒酵母ATCC204508纯培养物:挑取活化平板中酿酒酵母ATCC204508的单菌落接种至25 mL, YPD液体培养基中,在恒温震荡摇床中以30℃士1℃和180r/min的条件培养过夜得到种子液。取种子液0.5ml转接于10ml,YPD液体培养基中,在恒温震荡摇床中以30℃±1℃和180r/min的条件培养至OD为0.3~0.4(约2h~3h),得到酿酒酵母ATCC 204508纯培养物。
5.8植物乳杆菌CGMCC1.2437纯培养物:挑取活化平板中植物乳杆菌CGMCC1.2437的单菌落接种至25mLMRS液体培养基中,在恒温震荡摇床中以30℃士1℃和180r/min的条件培养过夜得到种子液。取种子液0.5ml转接于10mLMRS液体培养基中,在恒温震荡摇床中以30℃±1℃和180r/min的条件培养至OD为0.3~0.4(约2h~3h),得到植物乳杆菌CGMCC1.2437纯培养物。
6仪器设备
6.1微滴数字PCR 仪。
6.2核酸定量仪。
6.3热循环仪。
6.4电子天平:精度 0.01g。
6.5移液器:量程0.1μL~2.5μL,2μL~20μL,10μL~100μL和100μL~1 000 μL。
6.6恒温震荡摇床:温度可实现(28+1)℃和(37+1)℃,转速在125r/min~250 r/min。
6.7PH计:精度0.1.
6.8紫外-可见光分光光度计:可检测波长包含(600士20)nm,配备1cm比色皿。
7反应体系准备
7.1 酿酒酵母残留基因检测反应体系的准备
7.1.1 受试样品 DNA 模板准备
取1.00g受试样品,按照核酸提取试剂盒说明书进行DNA提取操作,最后使用50μL,的无菌进行DNA洗脱。提取得到的DNA样品使用核酸定量仪检测核酸的浓度和纯度。
7.1.2 阳性对照 DNA 模板准备
取植物乳杆菌纯培养物(5.8)100L,用无菌水稀释至OD为0.2(此时菌体浓度约为2X10CFU/mL)。采用梯度稀释的方法,继续用无菌水稀释至菌体浓度为1X10°CFU/mL。取100L稀释后的植物乳杆菌纯培养物,加入1.00g受试样品中作为阳性对照。利用DNA提取试剂盒对上述样品进行 DNA提取操作,最后使用50L的无菌水进行DNA洗脱。提取得到的DNA样品使用核酸定量仪检测核酸的浓度和纯度
7.1.3 阴性对照 DNA 模板准备
采用植物乳杆菌纯培养物(5.8)为阴性对照。利用DNA提取试剂盒对植物乳杆菌纯培养物进行DNA提取操作,最后使用50μL的无菌水进行DNA洗脱。提取得到的DNA样品使用核酸定量仪检测核酸的浓度和纯度
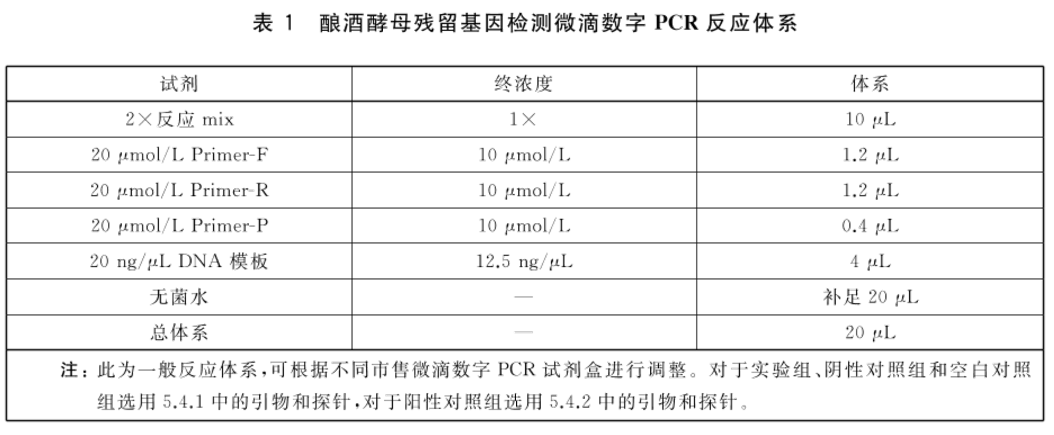
7.1.4微滴数字PCR 反应体系
每个试样的微滴数字PCR反应都设置3个平行,并按照表1设置PCR反应体系。用无菌水作为空白对照。
7.2 植物乳杆菌残留基因检测反应体系的准备
7.2.1 受试样品 DNA 模板准备
取1.00g受试样品,按照核酸提取试剂盒说明书进行DNA提取操作,最后使用50μL的无菌水进行DNA洗脱。提取得到的DNA样品使用核酸定量仪检测核酸的浓度和纯度。
7.2.2 阳性对照 DNA模板准备
取酿酒酵母纯培养物(5.7)100L,用无菌水稀释至D为0.2(此时菌体浓度约为2x10?CFU/ml),采用梯度稀释的方法,继续用无菌水稀释至菌体浓度为1X10”CFU/ml。取100L稀释后的酿酒酵母纯培养物,加人1.00g受试样品中作为阳性对照。利用DNA提取试剂盒对上述样品进行 DNA 提取操作,最后使用50μL,的无菌水进行DNA洗脱。提取得到的 DNA样品使用核酸定量仪检测核酸的浓度和纯度
7.2.3 阴性对照 DNA模板准备
采用酿酒酵母纯培养物(5.7)为阴性对照。利用DNA提取试剂盒对酿酒酵母纯培养物进行DNA提取操作,最后使用50μL,的无菌水进行 DNA洗脱。提取得到的DNA样品使用核酸定量仪检测核酸的浓度和纯度。
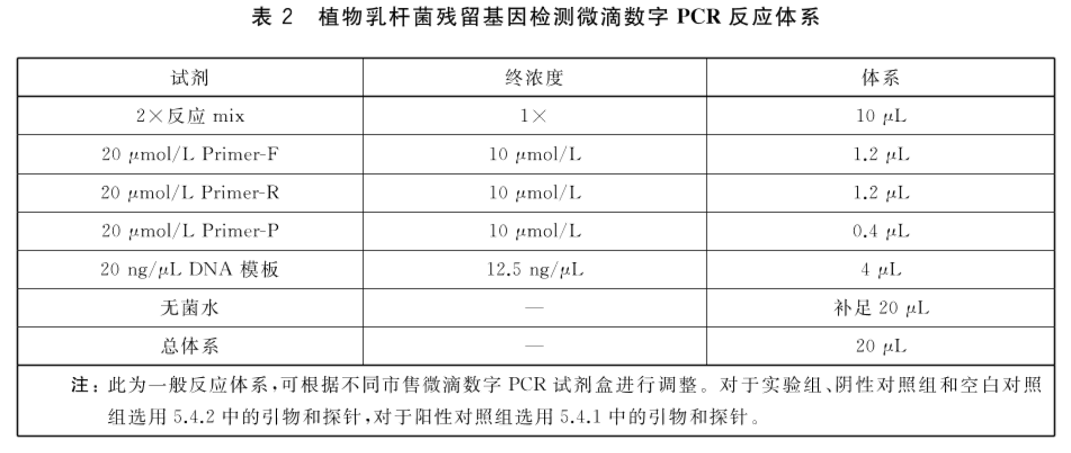
7.2.4 微滴数字 PCR 反应体系
每个试样的微滴数字PCR反应都设置3个平行,并按照表2设置PCR反应体系。用无菌水作为空白对照。
8微滴数字 PCR 反应
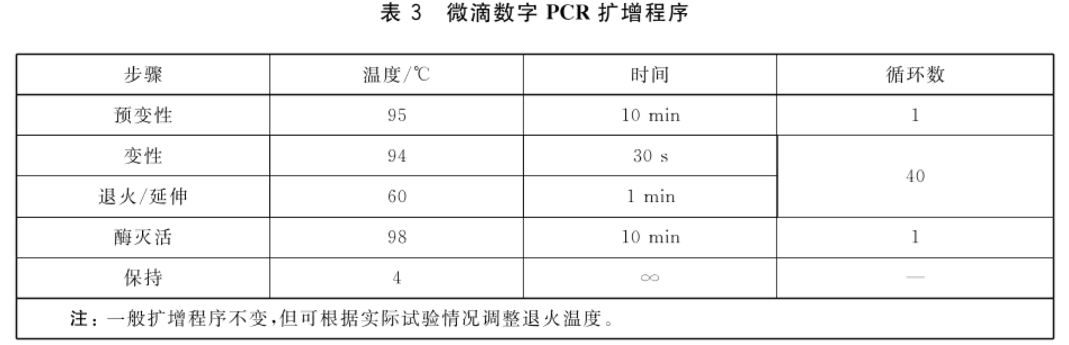
体系配置完成后按照数字PCR仪器的说明书进行操作,对反应体系进行分割获得油包水微滴。有效微滴数不应低于微滴数字PCR设备规定的微滴数量,否则需重复实验检测样本。按照微滴数字PCR仪的操作说明,在30min内进行PCR扩增,或者放于4℃冰箱4h之内进行PCR扩增。扩增可在数字PCR或热循环仪上完成,升降温速度≤2.5℃/s,扩增程序见表3。扩增结束后读取所有微滴的荧光信号,用于后续数据分析。
9结果分析与表述
9.1阈值设定
根据体系中阴性扩增体系终点荧光值设定荧光的阈限值。限值需明确区分扩增体系中的阴性微滴和阳性微滴。
9.2对照组检测结果分析
阴性对照组和空白对照中没有任何扩增现象,阳性对照组出现明显的扩增现象,表明此次微滴数字PCR法检测正常,无需重复检测,具有可信性.
9.3试样检测结果分析
试样检测的结果根据以下原则分析:
a)阳性对照和受试样本均出现明显的扩增现象,其扩增终点荧光信号值超过荧光阈限值,这表明试样中检测出目的微生物残留基因,表述为“试样中检测出目的微生物残留基因,检测结果为阳性”。
b)阳性对照得到了明显的扩增,其终点荧光信号值超过荧光阈限值,受试样本未出现明显的扩增现象,其扩增终点荧光信号值位于阈限值之下,这表明试样中未检测出目的微生物残留基因,表述为“试样中未检测出目的微生物残留基因,检测结果为阴性”
参考文献
《GB/T 38485-2021 微生物痕量基因残留测定微滴数字 PCR 法》
相关资源
名称:酿酒酵母|Saccharomyces cerevisiae
菌株编号:HZB300007
敬请关注“灰藻视界”,共筑健康未来!
— 武汉市灰藻生物科技有限公司团队敬上
灰藻生物:我们期待着与客户共同成长,共创生命科学的美好未来!
更新日期:2024-11-25
#创作团队编制人:木木
