CRISPR/Cas9的技术原理与sgRNA设计的关键要点
来源:武汉市灰藻生物科技有限公司 浏览量:7 发布时间:2026-02-03 16:48:56
引言
CRISPR/Cas9 技术作为近年来发展最迅速的基因编辑工具之一,已被广泛应用于基础研究、功能基因组学以及生物工程等多个领域。
相较于传统的基因操作方法,CRISPR/Cas9 具有设计灵活、操作相对简便以及适用范围广等优势,因此迅速成为分子生物学研究中的核心技术之一。
在 CRISPR/Cas9 体系中,引导 RNA(single guide RNA,sgRNA)的设计质量直接决定了基因编辑的效率和特异性。
本文将围绕 CRISPR/Cas9 的基本工作原理,重点介绍 sgRNA 的结构特点及设计过程中需要关注的关键要点,以帮助读者系统理解该技术的核心逻辑。
CRISPR/Cas9 技术概述
CRISPR(成簇规律间隔短回文重复序列)最初被发现于细菌和古菌的基因组中,是其抵御外源核酸入侵的一种适应性免疫系统。该系统能够记录曾经入侵的病毒或质粒序列,并在再次遭遇相同入侵时实现特异性识别与切割。
Cas9 是 CRISPR 系统中最常用的一种核酸内切酶,能够在特定位点对双链 DNA 进行切割。通过人工设计引导 RNA,研究人员可以将 Cas9 精确引导至目标基因组位置,从而实现定点基因修饰。
Cas9 蛋白的基本工作机制
Cas9 蛋白在引导 RNA 的辅助下识别目标 DNA 序列,并在靶位点产生 DNA 双链断裂。Cas9 的切割活性依赖于其两个关键催化结构域:HNH 结构域和 RuvC 结构域,二者分别负责切割 DNA 的两条互补链。
Cas9 识别靶序列时需要依赖 PAM(protospacer adjacent motif)序列的存在。以常用的化脓性链球菌来源 Cas9(SpCas9)为例,其识别的 PAM 序列通常为 5’-NGG-3’。缺乏合适 PAM 序列的 DNA 区域,即使与引导 RNA 完全互补,也不会被 Cas9 切割。
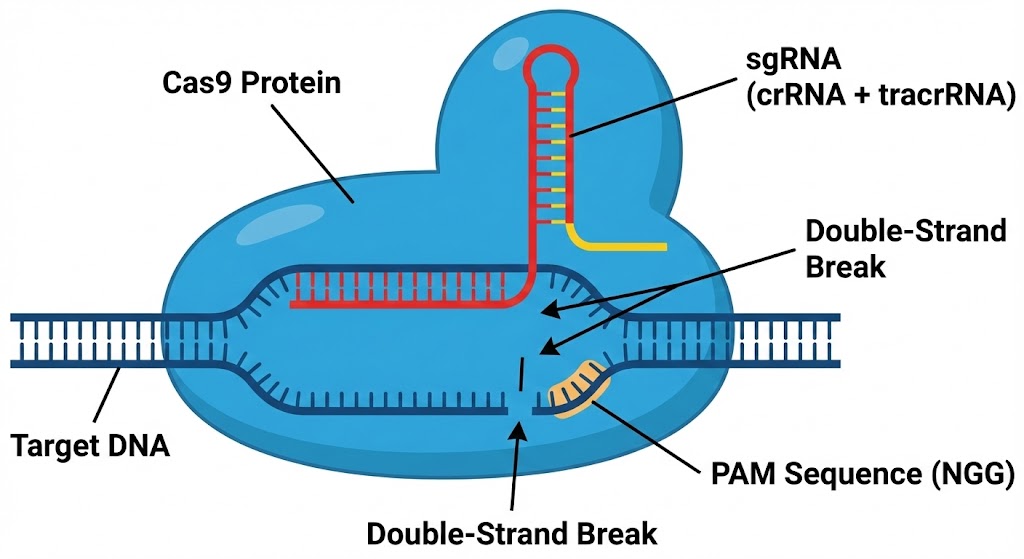
图1、CRISPR/Cas9 在 sgRNA 引导下识别目标 DNA 并产生双链断裂的基本机制示意图
不同 Cas 蛋白及 PAM 序列的差异
除常用的 SpCas9 外,目前已发现并开发出多种来源不同、特性各异的 Cas 蛋白。这些 Cas 蛋白在分子结构、切割方式以及对 PAM 序列的识别要求等方面均存在差异,从而拓展了 CRISPR 技术的应用范围。
例如,来源于金黄色葡萄球菌的 SaCas9 体积相对较小,更适合用于载体容量受限的递送系统;而其他来源的 Cas 蛋白则可能识别不同的 PAM 序列,使原本缺乏 NGG 位点的基因组区域也具备编辑可能性。
PAM 序列多样性的存在,为 CRISPR 实验设计提供了更高的灵活性。当目标区域缺乏特定 Cas 蛋白所需 PAM 时,合理选择其他 Cas 系统,往往能够为靶点设计提供新的解决思路。
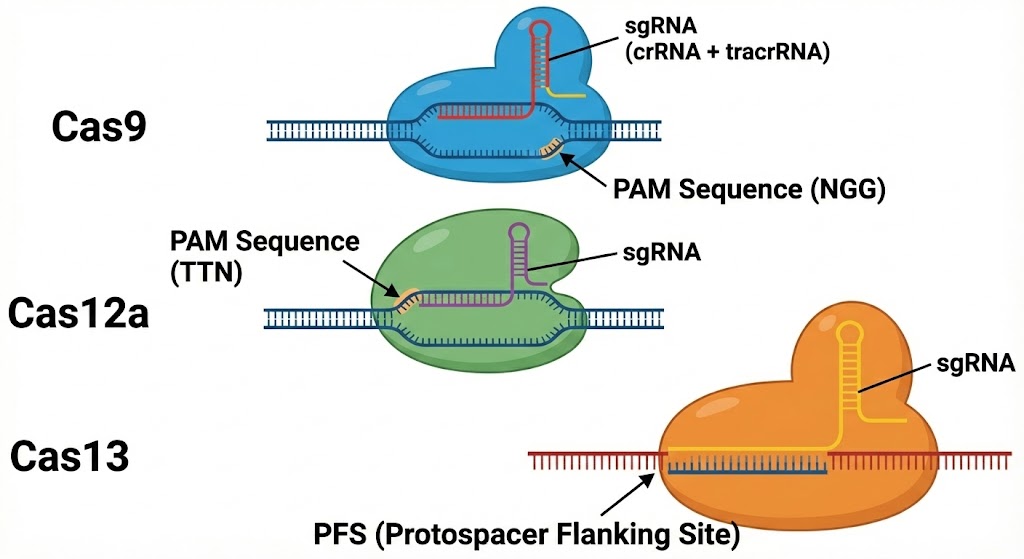
图2、不同 Cas 蛋白对 PAM 序列识别差异的示意图
什么是 sgRNA
在天然 CRISPR 系统中,Cas9 的定位依赖于两条 RNA 分子:crRNA 和 tracrRNA。crRNA 负责提供与目标 DNA 互补的序列信息,而 tracrRNA 则用于与 Cas9 蛋白结合并维持其结构稳定。
在实验应用中,这两条 RNA 被整合为一条单一 RNA 分子,即单链引导 RNA(single guide RNA,sgRNA)。sgRNA 同时承担靶序列识别和 Cas9 结合两种功能,大大简化了实验体系的设计。
sgRNA 通常由两部分组成:一段约 20 个核苷酸长度的靶向序列,以及一段相对固定的骨架序列。靶向序列决定了 Cas9 的切割位置,而骨架序列则确保 sgRNA 能够正确折叠并高效结合 Cas9 蛋白。
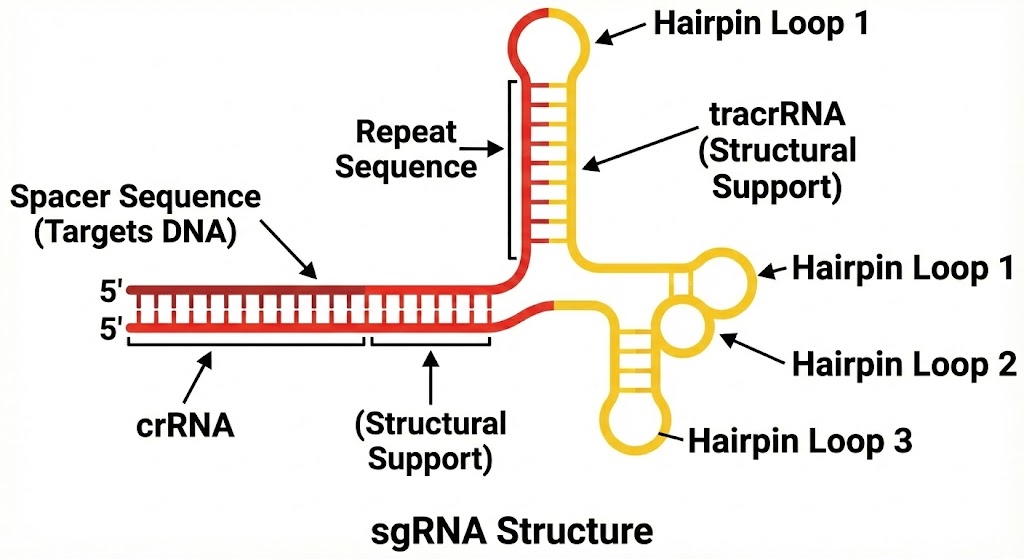
图3、单链引导 RNA(sgRNA)的结构组成示意图
sgRNA 设计的核心原则
sgRNA 的设计是 CRISPR/Cas9 实验成功的关键环节。理想的 sgRNA 应能够高效引导 Cas9 切割目标位点,同时尽量避免在基因组其他区域发生非特异性切割。
在选择 sgRNA 靶序列时,首先需要确认目标区域附近存在符合 Cas9 要求的 PAM 序列。对于 SpCas9,sgRNA 靶序列通常位于 PAM 上游的 20 个碱基。
此外,sgRNA 靶序列应尽量具有较高的基因组唯一性,避免与其他基因区域存在高度相似的序列。高度重复或同源性较强的区域,往往更容易产生脱靶效应。
sgRNA 靶序列的 GC 含量同样会影响编辑效果。过高或过低的 GC 含量都可能影响 sgRNA 与目标 DNA 的结合稳定性,一般建议选择 GC 含量适中的靶序列。
sgRNA 设计中常见的失败原因
在实际实验过程中,即使遵循基本设计原则,sgRNA 仍有可能表现出较低的编辑效率。这种情况往往并非单一因素导致,而是多种条件共同作用的结果。
首先,目标基因组区域的染色质状态会显著影响 Cas9-sgRNA 复合物的可及性。处于高度压缩状态的异染色质区域,往往不利于 Cas9 结合和切割。
其次,sgRNA 本身的二级结构也可能对其功能产生影响。某些靶向序列在转录后容易形成稳定的发卡结构,从而影响 sgRNA 与 Cas9 的结合效率或靶序列识别能力。
此外,不同细胞类型在 DNA 修复活性和 Cas9 表达水平方面存在差异,这也可能导致相同 sgRNA 在不同实验体系中表现出明显不同的编辑效果。
影响编辑效率与特异性的主要因素
CRISPR/Cas9 编辑效率和特异性不仅受 sgRNA 设计影响,还与 Cas9 蛋白类型、表达水平以及细胞自身的 DNA 修复机制密切相关。
在 DNA 双链断裂发生后,细胞通常通过非同源末端连接(NHEJ)或同源重组修复(HDR)途径进行修复。NHEJ 修复过程容易引入插入或缺失突变,因此常被用于基因敲除实验;而 HDR 则可在供体模板存在的情况下实现精确的序列插入或替换。
此外,不同 Cas9 变体在序列识别严格性方面存在差异。近年来开发的高保真 Cas9 变体,在降低脱靶效应方面表现出一定优势,但在部分实验体系中可能伴随编辑效率的变化。
CRISPR/Cas9 技术的典型应用场景
基于 CRISPR/Cas9 的基因编辑技术,目前已被广泛应用于多种研究场景。在基础研究领域,该技术常用于基因功能解析,通过构建基因敲除或敲入模型,研究特定基因在细胞或生物体中的作用机制。
在功能基因组学研究中,CRISPR 技术还可用于大规模筛选实验。通过构建 sgRNA 文库,研究人员可以在全基因组范围内系统评估不同基因对特定表型的影响。
此外,CRISPR/Cas9 技术在合成生物学、微生物工程以及生物技术产品开发等领域同样展现出广阔的应用前景,为相关研究和产业化探索提供了重要工具。
总结
CRISPR/Cas9 技术通过可编程的引导 RNA 实现对基因组的精准操作,而 sgRNA 的设计质量在其中起着决定性作用。深入理解 Cas9 的工作机制及 sgRNA 的设计原则,有助于提高基因编辑实验的成功率和结果可靠性。
随着相关工具和算法的不断完善,CRISPR/Cas9 技术将在生命科学研究和生物工程领域发挥更加重要的作用。
参考文献
1. Jinek M, Chylinski K, Fonfara I, Hauer M, Doudna J A, Charpentier E. A programmable dual-RNA–guided DNA endonuclease in adaptive bacterial immunity. Science, 2012.
2. Cong L, Ran F A, Cox D, et al. Multiplex genome engineering using CRISPR/Cas systems. Science, 2013.
3. Hsu P D, Lander E S, Zhang F. Development and applications of CRISPR-Cas9 for genome engineering. Cell, 2014.
4. Addgene. CRISPR Guide. https://www.addgene.org/crispr/
敬请关注灰藻生物,共筑健康未来!
— 武汉市灰藻生物科技有限公司团队敬上
更新日期:2026-01-30
编制人:小段
审稿人:小藻
