扩增子测序:原理、步骤、用途
来源:武汉市灰藻生物科技有限公司 浏览量:3490 发布时间:2024-11-01 20:47:17
扩增子测序介绍
扩增子测序是一种专门用于表征生物体内特定基因组区域或保守区域,并研究其遗传变异的测序方法。
与需要覆盖整个基因组的全基因组测序不同,扩增子测序采用聚合酶链式反应(PCR)技术,有选择性地扩增感兴趣的基因区域并进行测序。通过PCR,目标DNA区域得以放大,生成多个目标DNA的拷贝,即所谓的“扩增子”。
作为一种靶向测序技术,扩增子测序在识别遗传变异、探索微生物多样性以及检测临床样本中的病原体等方面有着广泛应用。由于该方法专注于特定区域,因此相比全基因组测序更为高效和精确,能够实现对特定基因区域的深入研究,而不必对整个基因组进行全面分析。
扩增子测序原理
扩增子测序的基本原理是利用PCR技术扩增特定的DNA区域,随后对这些扩增产物进行测序。
具体操作中,首先设计特异性寡核苷酸引物,用于扩增感兴趣的基因组区域。接着,通过PCR技术放大这些区域,生成短的、特异性的DNA片段——扩增子。之后,利用高通量测序技术对这些扩增子进行测序。这种方法的高度针对性确保了测序结果能够准确反映目标区域的特性,对于生物体的表征及遗传变异的检测具有很高的灵敏度。
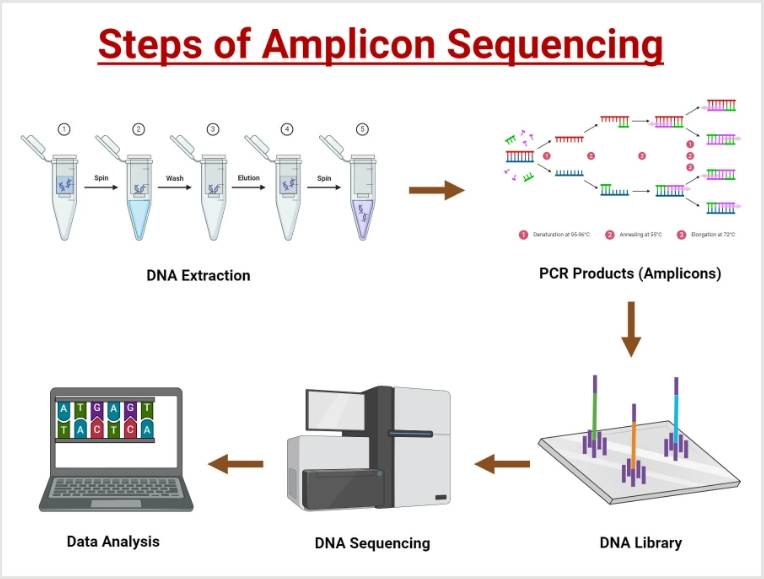
扩增子测序的过程大致分为几个步骤:首先从样品中提取感兴趣的遗传区域;然后通过PCR技术富集选定的目标区域;接下来添加接头以准备测序;形成文库后,可以将多个样本合并,在一次测序运行中同时处理,从而提高效率和经济性。
扩增子测序
扩增子测序的流程/步骤
扩增子测序步骤
样品制备和DNA提取
首先,从采集的样品中提取感兴趣的DNA。这一过程可以通过多种方法实现,如化学裂解法或机械破碎法等。目的是破坏细胞结构,释放出DNA,并将其与细胞残渣及其他杂质分离。经过一系列的纯化、沉淀和重新悬浮步骤,最终得到纯净的目标DNA。

DNA提取步骤
PCR扩增
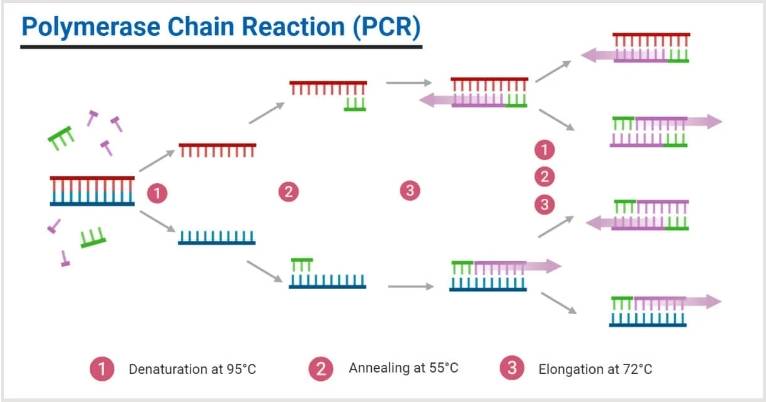
接下来,使用PCR技术扩增特定的DNA区域。这个过程包括多次循环的变性、退火和延伸步骤,以大量复制目标DNA区域。为此,需要设计针对特定基因组区域的特异性引物,这些引物不仅帮助定位并扩增目标区域,还会携带特定的条形码,以便在后续测序过程中区分来自不同样本的扩增子。PCR扩增后的产物需要进一步纯化,去除未参与反应的引物、引物二聚体、自由核苷酸等杂质,确保只有高质量的扩增子进入下一步。
PCR聚合酶链式反应
文库构建
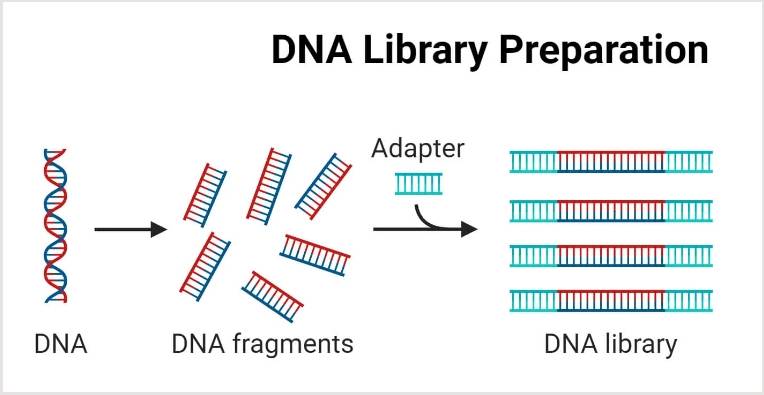
在扩增子上添加测序接头,构建用于测序的文库。这些接头是必需的,因为它们使扩增子能够固定在测序仪器的流动池上。添加接头后,需验证文库的质量,比如通过凝胶电泳检查扩增产物的大小分布和纯度,确认目标扩增子的存在。
基因文库构建
测序
准备好的文库会被加载到测序平台上进行测序。目前常用的测序平台有Illumina HiSeq、MiSeq以及PacBio等。其中,Illumina平台因其高通量特性而广泛应用于扩增子测序。HiSeq适用于大规模项目,尽管其处理时间较长;而MiSeq则更适合小规模项目,因为它更经济且快速。
数据分析 最后,对测序产生的原始数据进行生物信息学分析。这通常包括以下几个步骤:首先,利用条形码信息将不同样本的读数分开;接着,使用软件(如Trimmomatic)去除低质量碱基、适配器残留和污染物;随后,通过去重操作合并相同的序列并移除重复项;此外,还需执行嵌合体去除,以排除PCR扩增过程中可能产生的错误连接的DNA片段。之后,根据具体的研究目的,可以将序列聚类成操作分类单元(OTUs)或解析为高分辨率的扩增子序列变体(ASVs)。最终,通过与参考数据库对比,进行序列的分类学分配,并使用各种指标(如物种丰富度、均匀度、差异性和系统发育多样性)评估α多样性和β多样性,以全面了解样本间的微生物组成差异。这些分析通常借助于phyloseq、DESeq2等专业软件完成。
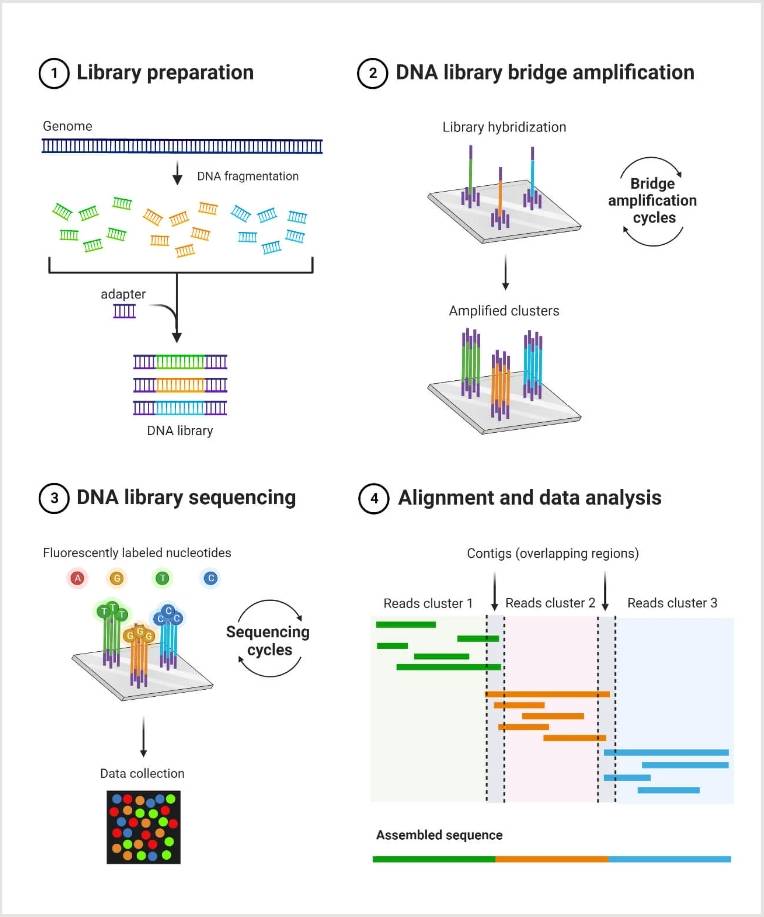
完整测序流程
扩增子测序的类型
根据测序目标的不同,扩增子测序主要可以分为以下三种类型:
16S rRNA测序
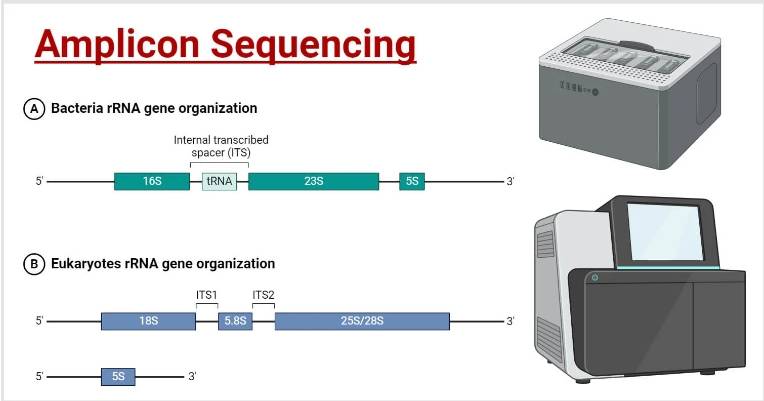
16S rRNA测序是指对存在于原核生物中的16S核糖体RNA(rRNA)基因进行测序。作为鉴定和研究细菌与古细菌最为常用的方法之一,16S rRNA基因由保守区和可变区组成。保守区为PCR扩增提供了稳定的引物结合位点,保证了扩增的准确性;而可变区则包含了各物种间独特的序列,有助于精确定位和分类不同类型的微生物。
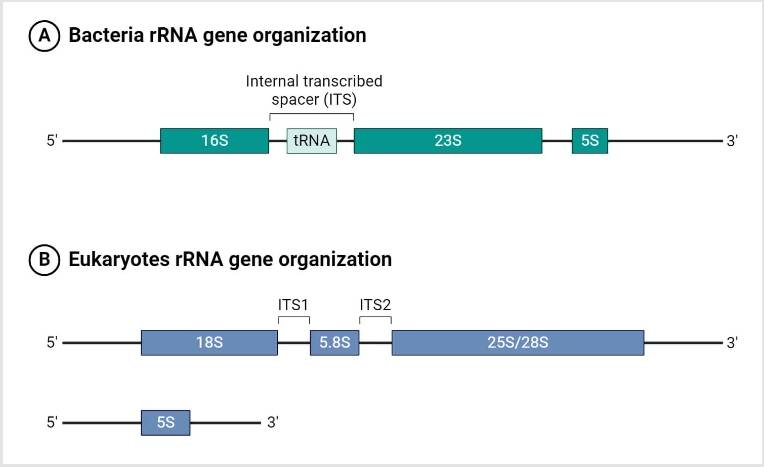
细菌和真核rRNA
18S rRNA测序
18S rRNA测序则聚焦于真核生物特有的18S rRNA基因,同样含有保守区和可变区。通过针对这一基因的测序,可以有效地识别和分类真核生物。保守区确保了引物的有效结合,而可变区则提供了区分不同真核生物种类的关键信息。
ITS测序
ITS测序专门针对真菌核糖体DNA中18S、5.8S和28S rRNA基因之间的内部转录间隔区(ITS)进行。该方法主要用于真菌的鉴定和分类学研究。ITS区域由于其高度的变异性,成为了区分亲缘关系接近的真菌物种的有效工具。
扩增子测序的优点
能够高效地识别和筛选遗传变异。
支持多目标区域的同时扩增,提高了目标区域的测序覆盖率。
相较于全基因组测序等方法,扩增子测序显著降低了测序成本和缩短了处理时间,因为所需读数较少,使用的试剂也更少。
对DNA输入的要求较低,适应范围广。
基于PCR的扩增子测序流程相对简单快捷,易于实施。
扩增子测序的局限性
使用PCR扩增可能会引入偏差,如非均匀扩增,这可能会影响测序结果的准确性。
不当的引物设计可能导致特异性下降或出现非特异性扩增。
该方法仅关注特定的基因区域,可能忽视了目标区域外的重要遗传变异。
当起始材料受到降解或污染时,可能会导致扩增效果不佳,产生错误的结果。
扩增子测序的应用
遗传疾病研究:用于检测与特定性状或遗传疾病相关的突变,特别是能够识别复杂样本中的罕见基因突变和低频突变。
病原体检测:在临床样本中识别和表征病原体,例如用于监测水体中的病原体分布及病毒变种的追踪。
农业生产:通过识别作物的遗传变异和检测植物病原体来管理农作物疾病。
系统发育学研究:通过对扩增子序列与现有数据库的比较,实现新物种的分类和识别,有助于研究不同物种间的遗传多样性和进化关系。
微生物生态学:用于识别和分类不同环境下的微生物群落。
法医学应用:在法医调查中进行DNA分析,辅助案件侦破。
参考资料
https://tuftsdatalab.github.io/Research_Technology_Bioinformatics/tutorials/AmpliconSeq/16STutorial.pdf
Bybee, S. M., Bracken-Grissom, H., Haynes, B. D., Hermansen, R. A., Byers, R. L., Clement, M. J., Udall, J. A., Wilcox, E. R., & Crandall, K. A. (2011). Targeted amplicon sequencing (TAS): a scalable next-gen approach to multilocus, multitaxa phylogenetics. Genome biology and evolution, 3, 1312–1323. https://doi.org/10.1093/gbe/evr106
Liu, Y. X., Qin, Y., Chen, T., Lu, M., Qian, X., Guo, X., & Bai, Y. (2021). A practical guide to amplicon and metagenomic analysis of microbiome data. Protein & cell, 12(5), 315–330. https://doi.org/10.1007/s13238-020-00724-8
敬请关注我们,共筑健康未来!
— 武汉市灰藻生物科技有限公司团队敬上
灰藻生物:我们期待着与客户共同成长,共同创造生命科学的未来!
更新日期:2024-11-01
#创作团队
编制人:小藻 | 审稿人:小灰
