菌株(细菌/真菌/古菌)鉴定服务
来源:灰藻科技 浏览量:2506 发布时间:2024-05-24 17:31:33
菌株鉴定介绍
菌株鉴定是指通过各种实验方法,确定微生物具体分类的过程,本服务主要是指基于DNA测序的分子生物学鉴定实验手段。微生物菌种DNA测序鉴定是一种利用DNA测序技术对微生物的物种进行准确识别的方法。它不依赖于微生物的形态、生理、生化等特征,而是通过分析微生物的核糖体RNA基因(rDNA)的序列,与数据库中的已知序列进行比对,从而确定微生物的生物学分类。微生物菌种DNA测序鉴定比传统的生化鉴定方法更快速、更准确、更全面,可以应用于医学、农业、环境、工业、食品等领域的微生物多样性分析。
Sanger测序技术
16S rDNA测序
16S rDNA测序技术是针对细菌和古细菌的一种鉴定方法,它利用16S rRNA基因的高变区作为标记,进行微生物的分类和鉴定。16S rDNA的长度约为1540个碱基对,大小适中,突变率小,是细菌系统分类学研究中常用、有用的“分子钟”,也被称为“细菌化石”,存在于所有原核生物的染色体基因组中。
16S rRNA基因既有恒定区(constant region),也有可变区(variable region),保守区反映了微生物的亲缘关系,可变区反映了微生物的差异。不同的高变区有不同的特异性和覆盖度,一般选择V3-V4区或V4-V5区进行测序。通过对16S rDNA进行PCR扩增和测序,可以获得原核生物的16S rRNA的序列信息,然后与数据库中的已知序列进行比对,从而确定原核生物的种类和相对丰度,目前被广泛用于医学、农业、环境等领域 。
18S rDNA测序
18S rDNA测序技术是针对真菌的一种方法,它利用18S rRNA基因的高变区作为标记,进行微生物的分类和鉴定。18S rRNA基因是编码真核生物核糖体小亚基rRNA的DNA序列,存在于所有真核生物的基因组中。
18S rRNA基因也有保守区和可变区,保守区反映了生物物种间的亲缘关系,可变区反映了物种间的差异。一般选择V4区或V9区进行测序,因为这些区域的特异性好,数据库信息全,分类效果好。18S rDNA测序技术可以鉴定出样本中的真核微生物的种类和相对丰度,目前被广泛用于水生生物、土壤生物、人体微生物等领域。
ITS测序
ITS测序是针对真菌的另一种方法,它利用内转录间隔区(Internally Transcribed Spacer,ITS)作为标记,进行微生物的分类和鉴定。ITS是位于真菌18S、5.8S和28S rRNA基因之间的非编码区,分别为ITS1和ITS2。
ITS由于承受较小的自然选择压力,在进化过程中能够容忍更多的变异,表现出极为广泛的序列多态性。ITS的保守型表现为种内相对一致,种间差异较明显,能够反映出种属间,甚至菌株间的差异。ITS序列片段较小(ITS 1和ITS 2长度分别为350 bp和400 bp),易于分析,目前已被广泛用于真菌不同种属的系统发育分析。
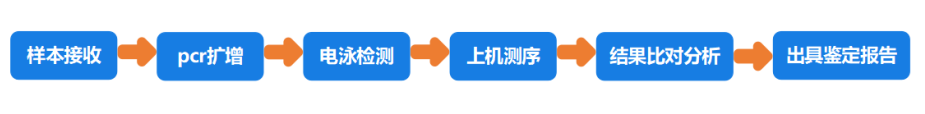
实验流程
菌株分子生物学鉴定的具体步骤如下:
- 样本准备:从新鲜培养物中挑选单一样本,通常是纯化菌落,清洗菌落以去除可能的污染;
- DNA提取:将菌落或菌液转移至离心管,使用试剂盒提取DNA,纯化DNA,检测DNA质量和浓度;
- PCR扩增:针对目标基因选择特异性引物,如16S rRNA(细菌/古菌)或18S rRNA/ITS区域(真菌),准备PCR反应体系(模板DNA、引物、dNTPs、Taq酶和缓冲液等),在热循环PCR仪上进行扩增(预变性、循环变性、退火和延伸等);
- PCR产物纯化:对PCR产物进行纯化,去除未结合引物、脱氧核苷三磷酸(dNTPs)和其他小分子杂质,常用凝胶电泳;
- DNA测序:将纯化的PCR产物,送入测序仪进行测序,常用Sanger测序技术;
- 序列分析:对序列数据进行质控、拼接和编辑,得到的完整rDNA序列,将其与NCBI的GenBank、EzBiocloud或其他微生物序列数据库进行比对,序列同源性统计分析;
- 鉴定报告:鉴定服务周期为2-4周,一般来说霉菌的鉴定周期较长,报告内容包括实验方法、测序结果、比对分析等。
- 实验流程
根据序列比对结果,按照相似度高低判断种属:
当相似度在99%以上,可判定二者种属相同;
当相似度在97%以上,可判定二者的属相同;
当相似度在95%以上,可判定二者的科相同。
信息登记
- 拉丁名称:以最新分类学名称为准
- 中文名称:广泛传播的中文名称
- 原始编号:
- 保藏编号:
- 生物安全等级:1级/2级/3级/4级
- 培养特性:营养需求,推荐培养基,最佳温度,培养时间等
- 需氧类型:专性好氧/兼性厌氧/微好氧/严格厌氧
- 备注说明:
送样要求
- 1、若寄送菌株样本,推荐低温运输以确保活性。建议纯化三次以上,确保平板和斜面为单一菌落;
- 2、请尽早向技术说明培养条件,若菌株对培养基有特殊要求,建议同时寄送适量培养基;
- 3、若寄送菌液形式样品,确保菌液浓度足够,离心后的菌体质量不少于0.5g;
- 4、若寄送菌株DNA样品,确保DNA浓度不低于10ng/μl,DNA总量不少于50μl;
- 5、若样品纯度或浓度不满足要求,进而导致测序结果不佳(如测序套峰),公司不承担相关责任;
- 6、革兰氏阳性菌细胞壁较厚,采用试剂盒常规方法较难破壁,建议直接提供DNA样品,减少测序周期;
- 7、实验室目前仅接受低风险样品(生物安全等级2级以下),安全等级2级以上微生物,请提供灭菌处理的DNA样品;
- 8、其它服务细节,请联系客服咨询。
常见问题
- Q:测序结果存在套峰的原因有哪些?
- A:在测序反应中,模板或引物异常都可能造成套峰现象,主要原因有以下几点:测序引物在模板上有两个结合位点形成套峰;模板不纯,如果是质粒或是菌液,原因是非单克隆,如果是PCR,原因为非特异性条带;模板序列的特殊结构,如poly结构、发卡结构等。其中,菌株不纯导致的套峰仍正常收费。
- Q:为什么测序结果找不到引物序列?
- A:如果是PCR样品,在测序报告上找不到引物序列是正常的。因为测序反应是通过对被荧光标记的ddNTP的读取而获得基因序列的,测序引物没有荧光标记,因此在测序结果中不会被识别。实际上不仅测序引物无法识别,受限于当前的测序技术,紧靠测序引物一侧的30~50个bp通常也无法确保识别。质粒或菌株样品使用通用引物测序,可能是因为PCR引物距离载体的通用引物位点过近,由于测序反应在距离起始位点30-50bp内的结果可信度不高,影响了正确读取PCR引物序列。>
敬请关注我们,共筑健康未来!
— 武汉市灰藻生物科技有限公司团队敬上
灰藻生物:我们期待着与客户共同成长,共同创造生命科学的未来!
2024.05.24
